pha1 0.09836 0.04560 2.157 0.0310 *
## beta1 0.79985 0.03470 23.052 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Log Likelihood:
## -169.8449 normalized: -0.8492246
##
## Description:
## Thu Nov 2 13:01:15 2017 by user:
对于 200 个观察,\(\beta\) 的估计是巨大的,标准差相对较小!
让我们深入探讨这一点。我在犹他大学数学系的超级计算机上进行了一些数值实验(译注:实际上,普通家用电脑也能应付)。下面是一个辅助函数,用于通过 garchFit()(在计算过程中屏蔽所有 garchFit() 的输出)来提取特定拟合的系数和标准差。
getFitData <- function(x,
cond.dist = "QMLE",
include.mean = FALSE,
...)
{
args <- list(...)
args$data = x
args$cond.dist = cond.dist
args$include.mean = include.mean
log <- capture.output(
{
fit <- do.call(garchFit, args = args)
})
res <- coef(fit)
res[paste0(names(fit@fit$se.coef), ".se")] <- fit@fit$se.coef
return(res)
}
第一个实验是在每个可能的截止点计算该特定序列的系数。
(在编写此文档时,不会评估以下代码块。我已将结果保存在 Rda 文件中。对于涉及并行计算的每个代码块都是如此。我在犹他大学数学系的超级计算机上执行了这些计算,在这里保存结果。)
library(doParallel)
set.seed(110117)
cl <- makeCluster(detectCores() - 1)
registerDoParallel(cl)
x <- garchSim(
garchSpec(
model = list(
alpha = 0.2, beta = 0.2, omega = 0.2)),
n.start = 1000, n = 1000)
params <- foreach(
t = 50:1000,
.combine = rbind,
.packages = c("fGarch")) %dopar%
{
getFitData(x[1:t])
}
rownames(params) <- 50:1000
下面我绘制这些系数,以及对应于两倍标准差的区域。该区域应大致对应 95% 的置信区间。
params_df <- as.data.frame(params)
params_df$t <- as.numeric(rownames(params))
ggplot(params_df) +
geom_line(
aes(x = t, y = beta1)) +
geom_hline(
yintercept = 0.2, color = "blue") +
geom_ribbon(
aes(x = t,
ymin = beta1 - 2 * beta1.se,
ymax = beta1 + 2 * beta1.se),
color = "grey", alpha = 0.5) +
ylab(expression(hat(beta))) +
scale_y_continuous(
breaks = c(0, 0.2, 0.25, 0.5, 1)) +
coord_cartesian(ylim = c(0, 1))

这是一幅令人震惊的画面(但不是我见过的最令人震惊的画面,这是最好的案例之一)。请注意,置信区间无法覆盖 \(\beta = 0.2\) 的真实值,直到大约 375 个数据点。这些间隔本应该在大约 95% 的时间内包含真实值!除此之外,置信区间相当大。
让我们看看其他参数的行为。
library(reshape2)
library(plyr)
library(dplyr)
param_reshape <- function(p)
{
p <- as.data.frame(p)
p$t <- as.integer(rownames(p))
pnew <- melt(
p, id.vars = "t", variable.name = "parameter")
pnew$parameter <- as.character(pnew$parameter)
pnew.se <- pnew[grepl("*.se", pnew$parameter), ]
pnew.se$parameter <- sub(".se", "", pnew.se$parameter)
names(pnew.se)[3] <- "se"
pnew <- pnew[!grepl("*.se", pnew$parameter), ]
return(
join(
pnew, pnew.se,
by = c("t", "parameter"),
type = "inner"))
}
ggp <- ggplot(
param_reshape(params),
aes(x = t, y = value)) +
geom_line() +
geom_ribbon(
aes(ymin = value - 2 * se,
ymax = value + 2 * se),
color = "grey",
alpha = 0.5) +
geom_hline(yintercept = 0.2, color = "blue") +
scale_y_continuous(
breaks = c(0, 0.2, 0.25, 0.5, 0.75, 1)) +
coord_cartesian(ylim = c(0, 1)) +
facet_grid(. ~ parameter)
print(ggp + ggtitle("NLMINB Optimization"))
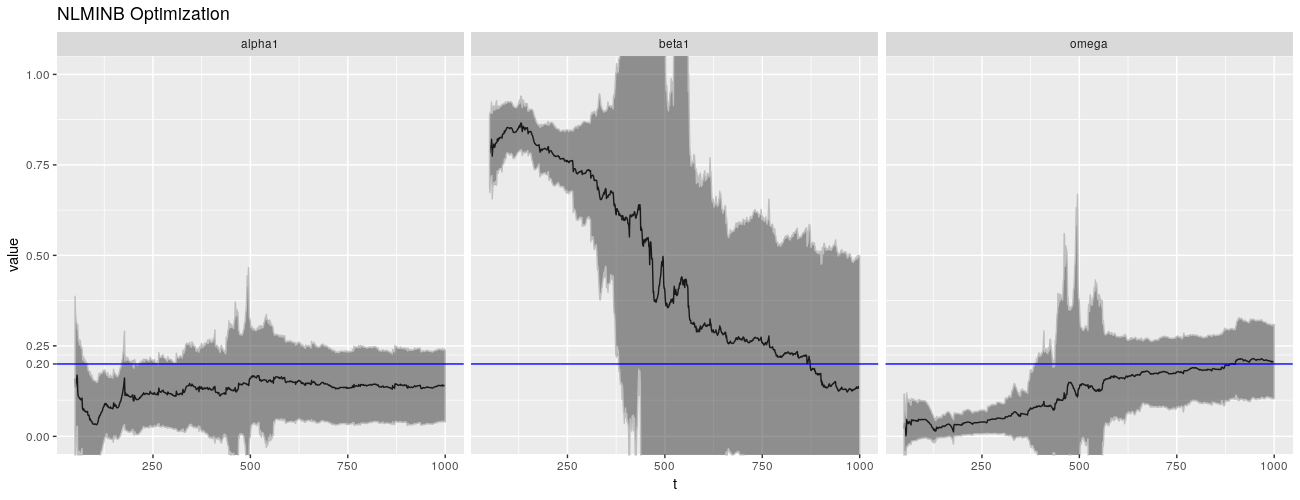
这种现象不仅限于 \(\beta\),\(\omega\) 也表现出不良行为。(\(\alpha\) 也不是很好,但要好得多。)
这种行为并不罕见,这是典型的。下面是使用不同种子生成的类似序列的图。
seeds <- c(
103117, 123456, 987654, 101010,
8675309, 81891, 222222, 999999, 110011)
experiments1 <- foreach(
s = seeds) %do%
{
set.seed(s)
x <- garchSim(
garchSpec(
model = list(
alpha = 0.2, beta = 0.2, omega = 0.2)),
n.star