在真实的世界中,缺失数据是经常出现的,并可能对分析的结果造成影响。在R中,经常使用VIM(Visualization and Imputation of Missing values)包来对缺失值进行可视化和插补。在使用VIM绘图时,有些绘图函数会对缺失值会自动进行插补。
缺失数据的分类:
- MCAR(完全随机缺失):若变量的缺失数据与其他任何观测或未观测的变量都不相关,则数据为MCAR.。
- MAR(随机缺失):若变量的缺失数据与其他观测变量相关,与未观测变量无关,则数据缺失是随机缺失。
- NMAR(非随机缺失):若缺失数据不属于MCAR和MAR,则数据是非随机缺失。
大部分处理缺失数据的方法都是假定数据是MCAR或MAR。
一,识别缺失值
R使用NA代表缺失值,NaN(不是一个数)代表不可能的值,符号Inf和-Inf分别代表正无穷和负无穷。
函数is.na()、is.nan()和is.infinite()分别用来识别缺失值,不可能值和无穷值。
函数complete.cases() 可以用来识别矩阵或数据框中的没有缺失值的行,若每行有一个或多个缺失值,则返回FALSE。注意,complete.cases()仅把NA和Nan识别为缺失值,无穷值(Inf和-Inf)被当作有效值。
二,探索缺失值的模式
在决定如何处理缺失数据前,了解哪些变量有缺失值、数目有多少、是什么组合等信息,是非常有用的。
1,列表显示缺失值
mice包中的md.pattern()函数可以生成一个以矩阵或数据框形式展示缺失值模式,0表示变量的列中存在缺失值,1则表示没有缺失值。注意,md.pattern()函数仅把NA识别为缺失值。
md.pattern(x, plot = TRUE)
例如,以VIM包提供的哺乳动物的睡眠数据(sleep,基础安装包中还有一个描述药效的sleep数据集)。
library(VIM) library(mice) data(sleep,package='VIM') md.pattern(sleep, false) BodyWgt BrainWgt Pred Exp Danger Sleep Span Gest Dream NonD 42 1 1 1 1 1 1 1 1 1 1 0 9 1 1 1 1 1 1 1 1 0 0 2 3 1 1 1 1 1 1 1 0 1 1 1 2 1 1 1 1 1 1 0 1 1 1 1 1 1 1 1 1 1 1 0 1 0 0 3 1 1 1 1 1 1 1 0 0 1 1 2 2 1 1 1 1 1 0 1 1 1 0 2 2 1 1 1 1 1 0 1 1 0 0 3 0 0 0 0 0 4 4 4 12 14 38
显示的结果中,模式是列的组合,第一列表示模式的数量,最后一列表示行中包含缺失值的列的数量,最后一行表示列中缺失值的数量。
比如,对于第一行,缺失值的数量是0,这样的数据行在数据集中有42个。对于第二行,包含缺失值的列(Dream和NonD 同时缺失)数量是2,这样的行在数据集中有9个。
2,用图形探究缺失数据
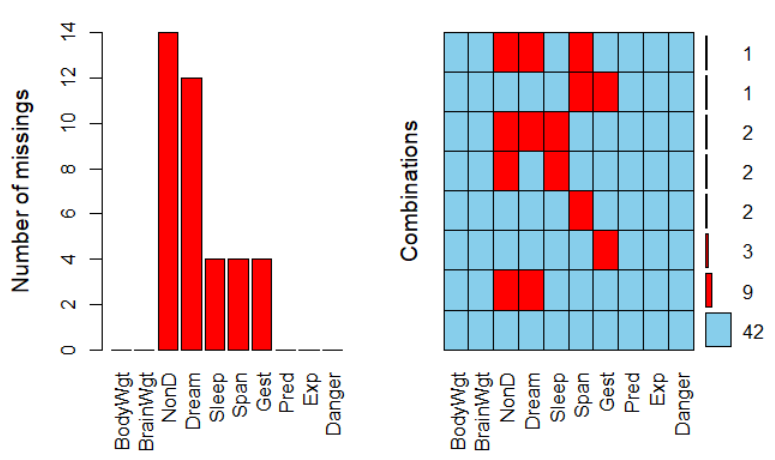
VIM包提供了大量能可视化缺失值模式的函数,aggr()、marginplot()和scattMiss()。
library(VIM) data(sleep,package='VIM') aggr(sleep,prop=FALSE,numbers=TRUE)
aggr重要参数注释:
- prob:当为TRUE时,显示为缺失值的占比;当为FALSE时,显示为缺失值的数量;
- numbers:是否显示数值,默认为FALSE,不显示缺失值的占比或数量。
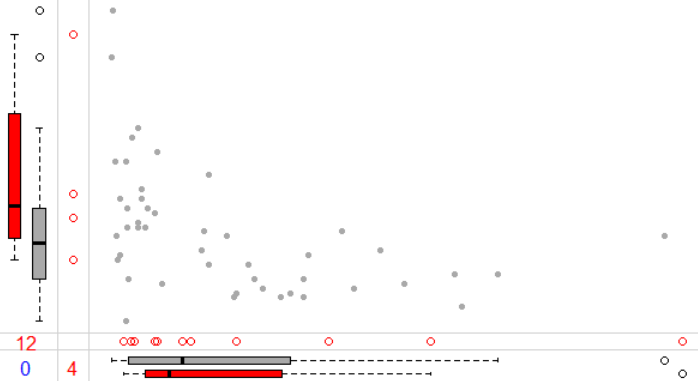
使用散点图来可视化两个变量的异常值:
library(VIM) data(sleep,package='VIM') marginplot(sleep[,c('Gest','Dream')],pch = c(20,1),col=c('darkgray','red','blue'))
对于包含两个变量的数据点,两个变量表示一个点(x,y),如果x和y都不包含缺失值,称作有效点;如果x或x中有一个包含缺失值,叫做单变量缺失,对于单变量缺失,该函数会自动插补;如果x和y都包含缺失值,叫做不可用点。
marginplot重要参数注释:
- x:包含两列的数据框,第一列是x轴,第二列是y轴,
- pch:点的形状,是个双值的向量,分别用于设置用于显示包含缺失值/插补值的点的形状,和有效点的形状。
- col:向量,用于设置绘图中使用的颜色。第一个颜色用于绘制有效点的散点图和箱图,第二个颜色用于绘制单变量缺失的散点图和箱图;第三个图颜色用于绘制不可用点。
红色箱图表示包含插补值之后的分布,灰色箱图表示有效点的分布。红色的空心点是插补之后的点。
3,用相关性探索缺失值
用指示变量代替数据集中的数据(1代表缺失,0代表存在),这样生成的矩阵有时被称作影子矩阵,求这些指示变量之间的相关性,有助于观察哪些变量经常一起缺失。
library(VIM) data(sleep,package='VIM') x <- as.data.frame(abs(is.na(sleep))) y <- x[which(apply(x,2,sum)>0)] cor(y) NonD Dream Sleep Span Gest NonD 1.00000000 0.90711474 0.48626454 0.01519577 -0.14182716 Dream 0.90711474 1.00000000 0.20370138 0.03752394 -0.12865350 Sleep 0.48626454 0.20370138 1.00000000 -0.06896552 -0.06896552 Span 0.01519577 0.03752394 -0.06896552 1.00000000 0.19827586 Gest -0.14182716 -0.12865350 -0.06896552 0.19827586 1.00000000
可以看到,Dream和NonD常常一起缺失(r=0.91)。
使用影子矩阵和原始数据之间的相关性,可以探索数据缺失的类型。
cor(sleep,y,use='pairwise.complete.obs') NonD Dream Sleep Span Gest BodyWgt 0.22682614 0.22259108 0.001684992 -0.05831706 -0.05396818 BrainWgt 0.17945923 0.16321105 0.007859438 -0.07921370 -0.07332961 NonD NA NA NA -0.04314514 -0.04553485 Dream -0.18895206 NA -0.188952059 0.11699247 0.22774685 Sleep -0.08023157 -0.08023157 NA 0.09638044 0.03976464 Span 0.08336361 0.05981377 0.005238852 NA -0.06527277 Gest 0.20239201 0.05140232 0.159701523 -0.17495305 NA Pred 0.04758438 -0.06834378 0.202462711 0.02313860 -0.20101655 Exp